实习日志2-cross-TCR interpreter
https://www.biorxiv.org/content/10.1101/2023.02.16.528799v2.full.pdf
此工作为阪大的蛋白质研究所成果,刚从Baker组回来的Koga也在这里吧
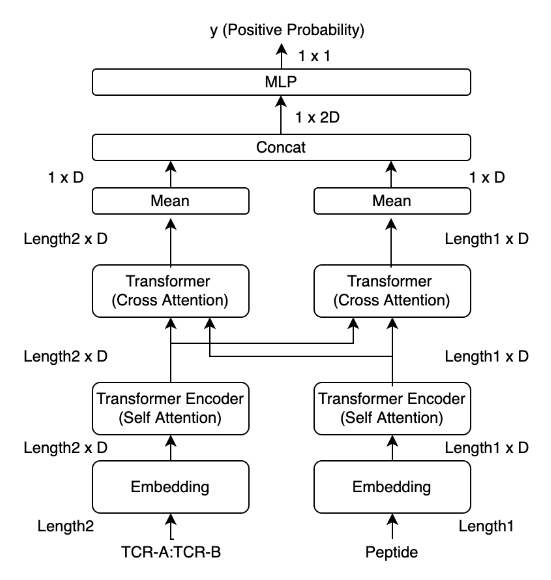
模型概述
在Concat前做了一次Cross Attention
复现测试部分
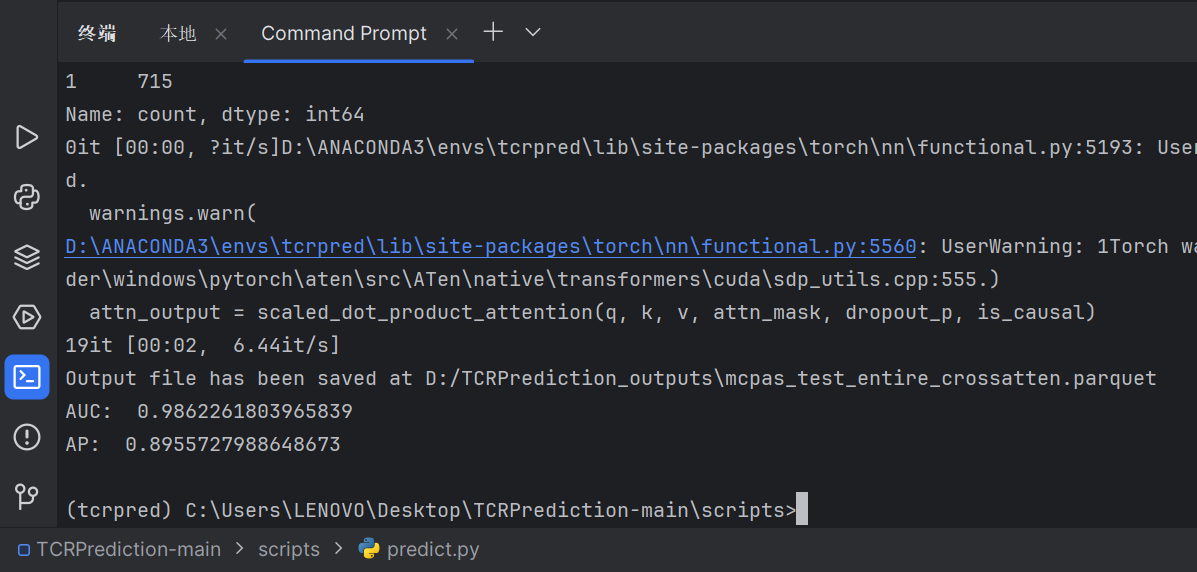
Mcpas_test上复现:
修改了输出文件地址 1
2
3
4
5
6
7
8# Update output filepath to D drive
output_dir = "D:/TCRPrediction_outputs"
if not os.path.exists(output_dir):
os.makedirs(output_dir)
base_name = pathlib.Path(args.input_filepath).stem
output_ext = pathlib.Path(args.input_filepath).suffix
output_filepath = os.path.join(output_dir, f"{base_name}_{args.model_key}{output_ext}")
修改checkpoints读取
1 | ckptpath = "C:/Users/LENOVO/Desktop/TCRPrediction-main/checkpoint/20230826_035401/best_20230826_035401_checkpoint_47_xent=-0.0421.pt" |
结果:
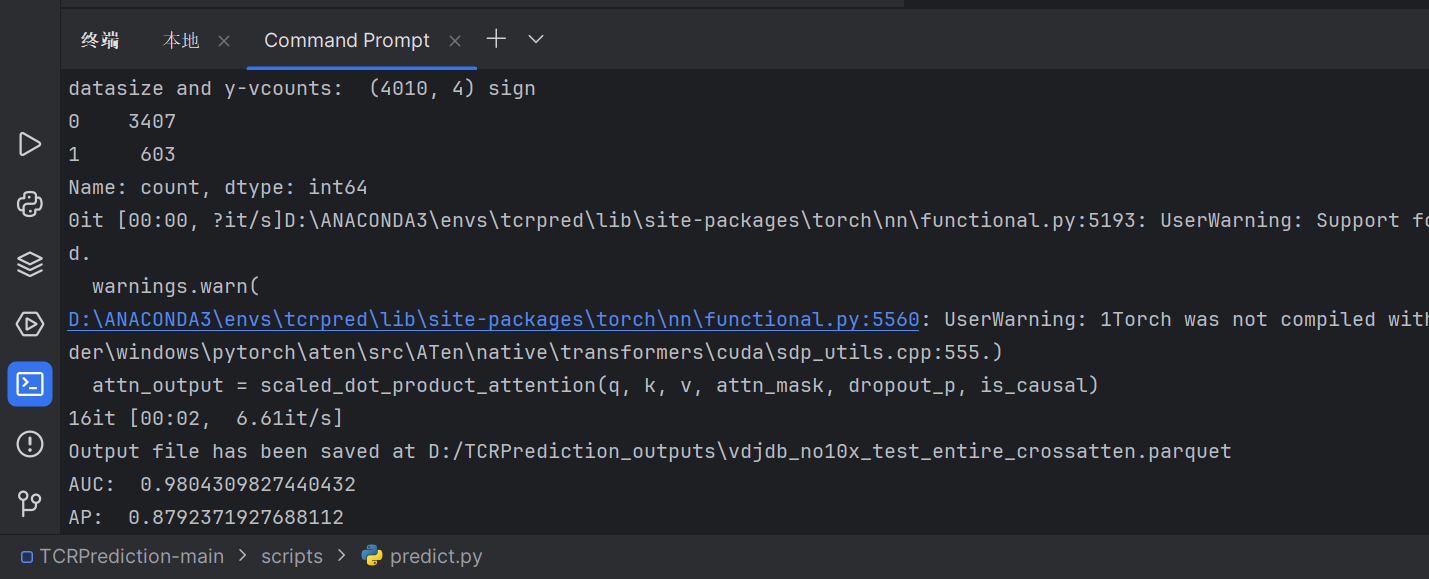
Vdjdb_no10x上复现:
结果:
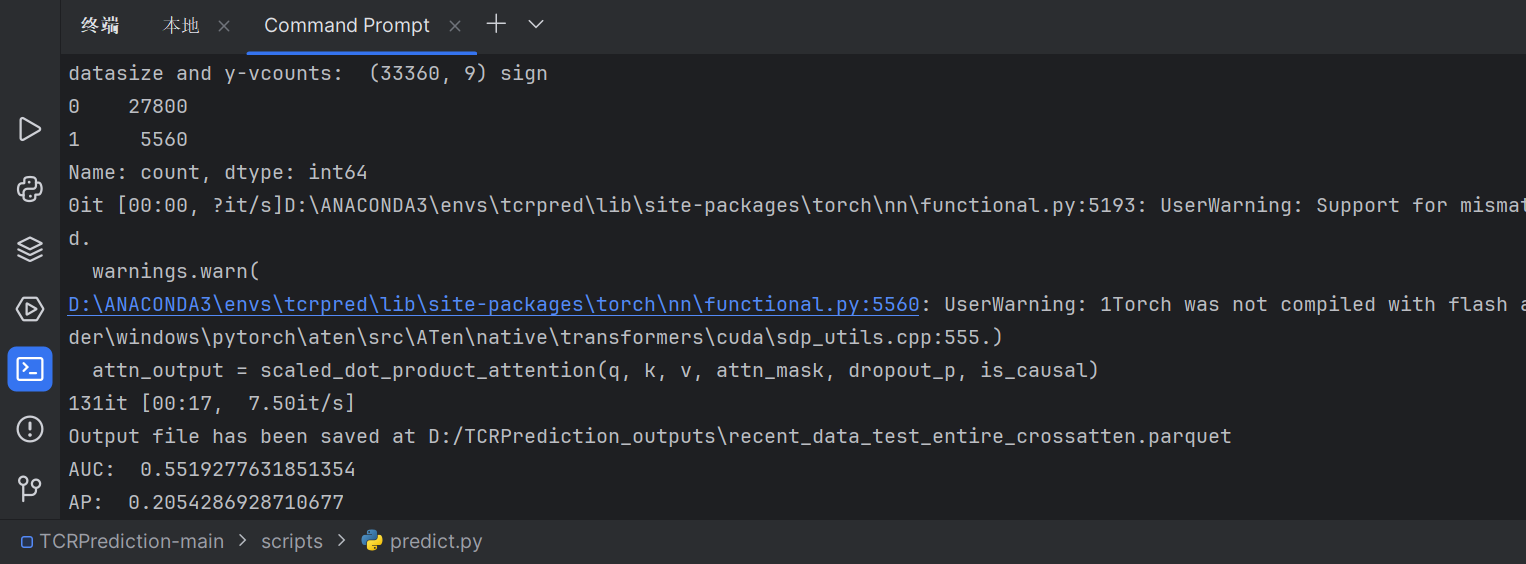
recent_data_test上复现:
结果:
本博客所有文章除特别声明外,均采用 CC BY-NC-SA 4.0 许可协议。转载请注明来自 Shouei的Blog!
评论