《LC-PLM》
ABSTRACT
近年来,自监督训练的语言模型在蛋白质序列的学习和生成性药物设计方面取得了巨大成功。许多蛋白质语言模型(protein LMs)基于Transformer架构,这些模型在蛋白质序列中提取出有意义的表示,并应用于下游任务。然而,Transformer模型的上下文长度有限,对于更长的蛋白质序列或蛋白质复合物,其表现相对较差。此外,Transformer在处理涉及蛋白质相互作用和生物网络中的动态机制时也存在局限性。为了克服这些问题,本文提出了一种新型的蛋白质语言模型,称为LC-PLM,基于BiMamba-S架构(选择性结构状态空间模型)来提升对长蛋白质序列的建模能力。LC-PLM采用了一种新的双向共享投影层设计,使其能够有效地捕捉氨基酸级别的细粒度信息。与此同时,LC-PLM的变体LC-PLM-G通过结合蛋白质-蛋白质相互作用(PPI)图,进一步在第二阶段的训练中加入生物学相互作用的上下文信息,提升了蛋白质结构和功能预测任务的表现。
INTRODUCTION
研究空白
- 尽管在DNA序列建模中已经证明SSM架构的潜力,但在蛋白质语言建模中的应用仍然有限。
- 如何利用这些架构的长上下文能力来建模蛋白质序列的图结构(如PPI图)尚未得到充分研究。
本研究的贡献
- 本文提出了LC-PLM(Long-context Protein Language Model)和其图上下文扩展模型LC-PLM-G,以克服上述问题。
- LC-PLM基于BiMamba-S架构,通过掩码语言建模(masked language modeling,MLM)学习蛋白质序列的高质量表示。
- LC-PLM-G通过随机游走策略,将PPI图上下文引入到蛋白质语言模型中,从而捕获生物相互作用信息。
创新点与总结
LC-PLM通过高效的SSM架构实现了: - 更好的神经扩展性(Neural Scaling Laws)。 - 更强的长度外推能力。 - 比基于Transformer的ESM-2模型在下游任务上性能提升7%-34%。
LC-PLM-G利用第二阶段的图上下文训练,在蛋白质功能预测、远程同源检测和PPI链接预测任务中表现出色。
研究展示了长上下文能力和生物交互信息的结合如何推动蛋白质语言建模的性能极限
LC-PLM: LONG CONTEXT PROTEIN LANGUAGE MODEL
先随机掩码,再embedding表示,进入BiMamba-S模块,再预测[Mask]处概率,计算交叉熵损失。
LC-PLM 是一种基于 BiMamba-S 架构的新型长上下文蛋白语言模型,旨在解决当前基于 Transformer 的蛋白语言模型在处理长序列或复杂图结构任务中的局限性。本文通过创新的架构设计和两阶段训练策略,显著提高了模型的长上下文建模能力和生物学任务表现。以下是详细介绍:
1. 模型架构设计:BiMamba-S
1.1 BiMamba 的基础设计
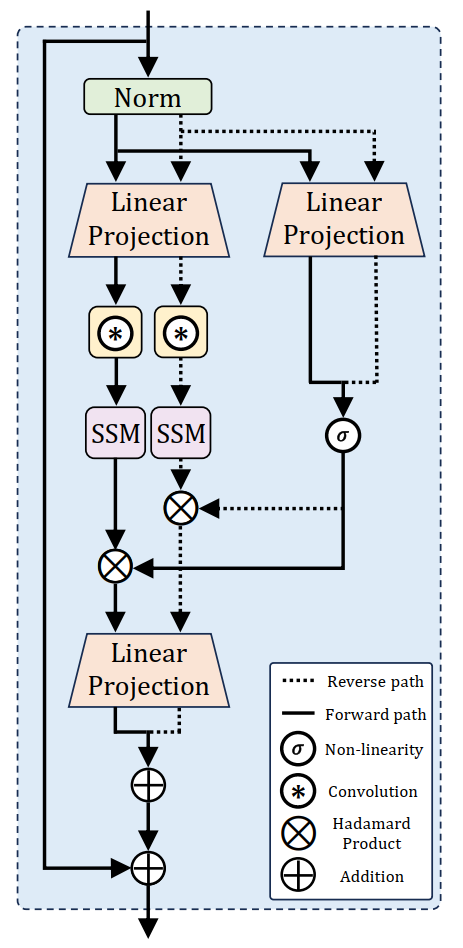
BiMamba 是 Mamba 模型的扩展,其架构基于选择性结构化状态空间模型(SSMs),专注于建模长序列数据。关键特性如下: - 捕获长距离依赖关系: - SSM 模块基于线性动态系统设计,能够有效捕获序列中的长距离依赖信息。 - 通过选择机制,在序列维度动态调整参数,实现复杂交互关系的建模。 - 双向扫描: - 输入序列从头到尾和从尾到头分别处理,结合正向和反向路径的信息,捕获全局上下文依赖。 - 特别适合掩码语言建模(MLM),能生成全面的序列表示。
1.2 BiMamba-S 的改进与优势
在 BiMamba 的基础上,LC-PLM 引入以下改进,形成 BiMamba-S: - 共享投影层(Shared Projection Layers): - 正向路径和反向路径共用线性投影层,使模型在几乎不增加参数量的情况下实现更大的深度。 - 共享投影层的设计降低了计算开销,同时提高了模型性能(评估损失降低 4.5%)。 - 未绑定输入和输出嵌入(Untied Input & Output Embeddings): - 输入嵌入和输出嵌入分离,避免传统方法中的各向异性问题。 - 提升了模型的表达能力和评估性能。
1.3 BiMamba-S 的工作流程
BiMamba-S 的主要步骤如下: 1. 输入处理: - 输入序列经过归一化并通过线性投影层生成特征表示。 - 特征分为正向路径和反向路径处理。 2. SSM 模块计算: - 正向路径特征通过标准 Mamba 模块处理,捕获序列正向信息。 - 反向路径特征通过翻转序列后同样输入 Mamba 模块处理。 - 正向和反向的输出通过残差连接与输入结合。 3. 预测头: - 最终输出经过归一化后输入预测头,用于生成掩码语言建模的预测概率分布。
1.4 BiMamba-S 的设计优势
- 长距离建模能力:
- 能高效捕获长序列中的长距离依赖关系。
- 参数效率:
- 共享投影层使得模型深度加倍但参数量保持不变。
- 全局上下文捕获:
- 双向扫描设计使模型捕获头尾信息,生成更全面的表示。
2. 双阶段训练策略
2.1 第一阶段:长上下文蛋白语言建模
- 目标:
- 通过掩码语言建模(MLM)学习通用且细粒度的蛋白质序列表示。
- 训练数据:
- 数据来源于 UniRef50 数据集,包含约 20B 条蛋白质序列。
- 掩码策略:
- 随机选择 15% 的氨基酸(AA)作为目标掩码。
- 80% 替换为
[MASK]。 - 10% 替换为随机 AA。
- 10% 保持不变。
- 80% 替换为
- 随机选择 15% 的氨基酸(AA)作为目标掩码。
- 特点:
- BiMamba-S 的 SSM 模块高效捕获长距离依赖。
- 生成的表示在蛋白质家族、功能和结构等任务中通用。
2.2 第二阶段:图上下文蛋白语言建模
- 目标:
- 捕获蛋白质之间的交互信息,将模型优化为任务相关表示。
- 输入构建:
- 随机游走生成序列:
- 在 PPI 图上进行随机游走,将每次游走的节点连接生成多蛋白序列。
- 正样本由随机游走路径生成,负样本由断开的节点对生成。
- 特殊标记设计:
[BON]和[EON]分别标记节点的开始和结束。[EDGE]和[NO_EDGE]表示节点间是否存在边。
- 随机游走生成序列:
- 训练策略:
- 在 MLM 的基础上引入图拓扑信息,通过特殊标记优化模型。
3. 设计选择的创新性与优势
3.1 BiMamba-S 的优势
- 优越的扩展性:
- 在处理超长序列时,评估损失显著低于基于 Transformer 的模型。
- 参数高效性:
- 共享投影层的设计使模型在保持相同参数量的情况下实现更深网络架构。
- 多任务适配性:
- LC-PLM 可适配多种任务(如蛋白质功能预测和结构预测)。
3.2 图上下文的增强
- 捕获生物学交互信息:
- 第二阶段通过 PPI 图增强,使模型更好理解蛋白质间的交互关系。
- 通用性与专用性结合:
- 第一阶段生成通用表示,第二阶段调整为任务相关的专用表示。
总结
LC-PLM 通过 BiMamba-S 实现了高效的长上下文蛋白语言建模,并结合双阶段训练策略,既能生成通用的蛋白质序列表示,又能捕获生物学交互信息。其创新性设计在蛋白质建模领域具有重要实践价值。