《论文复现:SenseXAMP》
Fuse feeds as one: cross-modal framework for general identification of AMPs
摘要
在这项研究中,我们提出了SenseXAMP,一个跨模态框架,通过利用输入序列的语义嵌入和蛋白质描述符(PDs)来提高AMPs识别性能。SenseXAMP包含一个多输入对齐模块和跨表示融合模块,以探索两种输入特征之间的隐藏信息,更好地利用融合特征。
为更好地解决AMPs识别任务,我们累积了最新的标注AMPs数据,形成了更大规模的基准数据集。此外,我们通过增加AMPs回归任务来扩展现有的AMPs识别任务设置,以满足抗菌活性预测等更具体的需求。实验结果表明,SenseXAMP在包括常用AMPs分类数据集和我们提出的基准数据集在内的多个AMPs相关数据集上,均优于现有的最先进模型。此外,我们进行了系列实验,证明传统PDs和蛋白质预训练模型在AMPs任务中的互补性。我们的实验结果显示,SenseXAMP可以有效结合PDs的优势,提高蛋白质预训练模型在AMPs任务中的性能。
介绍与前人工作概述
传统机器学习方法
- 机器学习方法的应用:近年来,基于机器学习的方法已成为计算识别抗菌肽(AMPs)的主流。这些方法通常依赖于蛋白质描述符(PDs)作为输入,这些描述符通过计算工具捕获蛋白质序列的物理、化学和组成特性。
- 常用的分类器:传统的机器学习方法中,常用的分类器包括支持向量机(SVM)、随机森林(RF)、极端梯度提升(Xgboost)和模糊K近邻(fuzzy KNN)。
- 已开发的流行方法:一些基于机器学习的流行方法包括AntiBP、AntiBP2、CAMP、IAMPE和ClassAMP。这些方法利用一种或多种传统机器学习算法,建立了AMPs识别模型。
深度学习方法
- 特征工程简化:深度学习方法简化了特征工程过程,近年来深度神经网络(DNN)模型已成为AMPs识别任务的主导方法。
- 基于卷积神经网络(CNN)的方法:例如,APIN、DeepAmPEP30和GRAMPA利用CNN作为识别AMPs的骨干网络。
- 基于双向长短期记忆(Bi-LSTM)的方法:如AMPScanner和Deep-ABPpred,使用Bi-LSTM构建序列模型。
- 基于BERT微调的方法:如AMP-BERT,随着自然语言处理研究的进展,基于大规模序列数据库预训练的蛋白质语言模型在多种生物信息学任务中显示出了巨大潜力,包括蛋白质结构预测和功能注释。最新的模型之一是ESM-1b(Evolutionary Scale Modeling),基于Transformer架构并在Uniref50上训练。
传统PDs的潜力
- 对传统PDs的再评估:尽管深度学习模型在AMPs任务中显示出色,但传统PDs在AMPs识别任务中的巨大潜力仍然值得探索。García-Jacas等人进行了一项综合评估,比较了仅使用传统特征描述符训练的浅层模型和DNN模型在AMPs预测任务中的表现。实验结果表明,浅层模型在某些情况下具有优势,至少与深度学习模型相当,这与之前基于小规模数据集得出的结论相反。
- 传统PDs的应用实例:例如,SMEP使用传统PDs进行大规模AMP筛选的湿实验结果进一步展示了传统特征描述符在AMP预测任务中的巨大潜力。
数据集的规模和平衡问题
- 小规模和平衡数据集的局限性:大多数AMPs识别研究通常使用相对较小且平衡的数据集,单一目标为AMPs分类。然而,在实际场景中,大多数来自自然来源的候选序列不是功能性AMPs。在这种情况下,训练在平衡数据集上的模型可能在实际应用中会出现过拟合问题。
- 增加回归任务的必要性:在候选库较大且用户对肽的抗菌活性有具体要求的情况下,仅进行分类任务可能不足。因此,研究如何从大规模、不平衡的候选库中识别潜在的AMPs,并考虑抗菌活性的具体要求,是必要的。
综合任务和模型融合
- 综合AMPs筛选任务:本研究提出了一种更具代表性的AMPs筛选任务组合,结合传统的AMPs分类任务和AMPs回归任务。相关研究表明,回归任务可以通过预测MIC值来辅助AMPs的细粒度筛选。
- 模型融合的局限性和创新:在对基于传统PDs的模型和预训练模型的错误样本分析中,发现两者的错误样本有很大比例不重叠,难以通过简单的模型融合技术(如堆叠法)纠正所有错误样本。因此,作者提出SenseXAMP模型,通过在特征级别上融合蛋白质预训练模型和传统PDs,显著提高了AMPs任务的性能。
总结
前人的工作展示了基于传统机器学习和深度学习方法在AMPs识别任务中的成功应用,尤其是在特征提取和模型构建方面。然而,这些方法在处理大规模、不平衡数据集和复杂任务组合方面仍存在局限性。SenseXAMP通过在特征级别融合传统PDs和预训练模型的信息,克服了这些局限性,并在多个AMPs相关数据集上展示了卓越的性能。
数据集
论文的“数据集”部分详细介绍了用于AMPs筛选任务的数据集构建过程以及这些数据集的组成部分。以下是该部分的详细描述:
数据集构建背景
AMPs筛选的实际场景中存在样本不平衡的情况,其中在大量候选肽中只有少量具有抗菌活性。为了构建数据分布更接近实际AMPs筛选场景的数据集,作者整合并清理了现有的AMPs识别相关数据集,形成了多个更大、更综合的AMP数据集。此外,考虑到更精确预测AMPs抗菌活性的重要性,作者还包括了AMPs回归数据集。
数据集组成
数据集主要由以下几部分组成:
- AMPs分类数据集:
- 平衡数据集:用于AMPs二分类任务。正负样本的长度和数量一致。
- 不平衡数据集:更接近现实情况,正负样本之间存在不平衡,挑战更大。
- AMPs回归数据集:主要用于预测AMPs的抗菌活性,以最低抑菌浓度(MIC)表示。选择了常用的两种细菌菌株作为目标菌株:
- 大肠杆菌(E. coli)数据集:主要由对大肠杆菌具有抗菌活性的AMPs和一批非AMPs组成。
- 金黄色葡萄球菌(S. aureus)数据集:主要由对金黄色葡萄球菌具有抗菌活性的AMPs和一批非AMPs组成。
数据集构建过程
- 数据收集和清理:
- 收集了11个前人工作的AMP相关数据集。这些序列来自多个知名的公开数据库,如抗菌肽数据库(APD)、抗菌肽数据存储库(DRAMP)、YADAMP(Yet Another Database of AMPs)、CAMP(Collection of AMPs)和DBAASP(Database of AMPs with Mainly Synthetic Peptides)。GRAMPA数据集提供了AMPs回归数据集的重要信息。
- 删除重复序列后,获得总计35,016个AMPs和175,341个非AMPs。
- 数据清洗:
- 删除包含特殊氨基酸(B、J、O、U、X和Z)的序列,仅保留长度在6到50之间的序列,因为较长的肽可能不稳定且有毒,而较短的肽不太可能具有抗菌活性。
- 对于不同数据集中标签不一致的序列,视为数据噪声并删除。
- 相似序列去重:
- 使用CD-HIT程序对正样本和负样本分别进行处理,去除高度相似的序列(相似度阈值为0.7)。处理后,正样本和负样本中不再存在超过70%相似度的序列。最终得到7,941个AMPs和28,021个非AMPs,用于创建不平衡分类数据集。
- 随机从28,021个非AMPs中选择7,941个,与AMPs的长度分布一致,创建正负比为1:1的平衡分类数据集。
- 回归数据集构建:
- 从GRAMPA数据集中选择目标菌株为大肠杆菌和金黄色葡萄球菌的数据,保留仅包含“C-末端:AMD”注释且序列长度大于5的序列。
- 对于同一肽的多个实验条件下的MIC值,取平均值作为最终标签。
- 大肠杆菌回归数据集中包含1,724个正样本和8,620个负样本,负样本从经CD-HIT过滤的非AMPs数据集中随机采样,正负比为1:5,以模拟实际场景。
- 金黄色葡萄球菌回归数据集类似地包含1,678个正样本和8,390个负样本。
- 标签设置与GRAMPA和SMEP数据集一致,所有非AMPs的MIC值设为log10(8196) ≈ 3.91,因此所有AMPs的标签小于此值。
数据集概览图
论文中的图1展示了数据集形成的流程,详细描述了数据收集、清洗、去重和构建分类及回归数据集的步骤。
)
在本研究中,我们提出了一种用于AMPs识别任务的全新端到端方法,名为SenseXAMP。该方法包含三个主要组件:
方法概述
SenseXAMP由以下三个主要模块组成: 1. 特征提取模块:将序列输入提取为两种模态的特征,包括蛋白质预训练模型和传统PDs计算工具。 2. 多输入对齐模块:提取并压缩两种模态特征的维度,以便进行有效融合。 3. 跨表示融合模块:结合两种模态的隐藏信息,以获得更全面的输入序列表示。
特征提取模块
在特征提取模块中,使用自监督蛋白质预训练模型和传统的PDs计算工具提取两种模态的特征,如图2a所示。自监督蛋白质预训练模型使用ESM-1b Transformer将序列输入转换为氨基酸残基级别的嵌入。每个序列的嵌入维度为 ([ , ]),其中 () 是序列的长度,() 对于ESM-1b是1280。我们还使用计算工具计算输入序列的伪氨基酸组成(PseAAC)、组成/转移/分布(CTD)等物理化学特征。该工具能够为给定输入序列生成676个物理化学特征。这些特征作为氨基酸残基级别嵌入的补充信息。
多输入对齐模块
在获得序列的两种模态特征后,SenseXAMP的多输入对齐模块进一步提取并对齐两种模态特征的维度,以进行后续的融合过程。如图2b所示,我们使用自注意力块从ESM-1b获得的嵌入中提取特征。该模块类似于Transformer编码器,由N个相同的层堆叠而成。每个层由多头自注意力和前馈神经网络(FFN)组成,每个子层都有残差连接。形式上,我们将输入序列嵌入表示为 (E(x) ^{N D}),其中N是序列中的标记数,每个标记的嵌入维度为D(这里为1280)。
多头自注意力是该模块的核心,它是缩放点积注意力的改进版。具体来说,输入序列嵌入的表示为 (E(x)),其计算公式如下:
[ Q = E(x)W_q, K = E(x)W_k, V = E(x)W_v ] [ (Q, K, V) = ()V ] [ (Q, K, V) = (_1, , _h)W_O ]
自注意力块的输出表示为 (F_{}),与输入序列嵌入的维度相同。
对于通过PDs计算工具计算的PDs特征,特征提取器由若干层全连接网络(FCN)组成,用于将其维度映射到与嵌入相同的维度D。形式上,我们将映射后的PDs特征表示为 (F_{} ^D)。
跨表示融合模块
SenseXAMP的跨表示融合模块起到了关键作用。多输入对齐模块进一步提取特征后,特征的维度已经对齐,可以进行特征级别的融合。如图2c所示,跨表示融合模块的核心类似于自注意力块,由多头注意力和FFN组成。需要注意的是,Q是从 (F_{}) 计算的,而K和V是从 (F_{}) 计算的。在此过程中,融合特征不断更新,而PDs特征保持不变。通过有效地整合和融合多种模态的信息,该模块可以提高分类和回归模型的准确性和鲁棒性。
损失函数
跨表示融合模块获得的最终融合特征具有强大的表示能力,通过输入到最终任务特定的头部(如回归、分类等)进行输出预测。在模型训练过程中,从多输入对齐模块获得的 (F_{}) 和 (F_{}) 特征,以及最终融合特征,分别输入到预测头部以获得相应的预测结果,然后用于计算损失函数。为了提高特征对齐过程中嵌入特征和PDs特征的提取性能,我们认为有必要监督每个模态特征的预测结果。通过简单地修改模型末端使用的预测头部,SenseXAMP可以方便地适应多种任务,如分类、回归等。在本研究中,我们使用二元交叉熵(BCE)作为AMPs分类任务的损失函数,对于AMPs回归任务,我们使用均方误差(MSE)作为损失函数:
[ (y, ) = - {i=1}^{n} ] [ (y, ) = {i=1}^{n} (y_i - _i)^2 ]
最终损失是所有头部预测损失的组合:
[ L = r_1 L_1 + r_2 L_2 + r_3 L_3 ]
其中 (r_1, r_2) 和 (r_3) 是权衡各损失项对整体损失贡献的可调超参数。
模型性能评估指标
对于AMPs分类任务,我们使用五个量化指标评估模型性能:准确率(ACC)、特异性(Sp)、敏感性(Sn)、马修斯相关系数(MCC)和F1分数。在实际场景中,非AMPs数量远多于AMPs,研究人员通常希望尽可能筛选出所有潜在的AMPs。因此,在这些评价指标中,综合评价指标如F1分数、MCC和反映正样本识别准确性的指标如Sn更为重要。
[ = ] [ = ] [ = ] [ = ] [ = ]
对于AMPs回归任务,我们主要使用topK-mse评估模型性能。序列的MIC值越小,抗菌性能越好。我们选择多个尺度的MSE全面评估模型性能,包括top10-mse、top30-mse、top100-mse、pos-mse和mse。topK-mse评估模型对具有最佳抗菌性能的前K个序列的MIC预测误差,pos-mse评估所有AMPs的MIC预测误差:
[ = _{i=1}^{K} ( i - y_i )^2 ] [ = {i=1}^{N} ( _i - y_i )^2 ]
其中K是选择评估性能的最佳抗菌性能序列数,N代表数据集的总AMPs数。
在AMPs筛选的背景下,非AMPs数量远多于AMPs,AMPs回归数据集模拟了这种不平衡的样本场景,这意味着如果模型预测所有序列的MIC为
(_{}),则可以获得足够低的MSE。然而,我们的最终目标是识别具有良好抗菌活性的AMPs,因此高性能模型不仅应关注MSE,还应关注反映AMPs(正样本)抗菌活性预测质量的指标,如pos-mse和topK-mse。这并不意味着模型仅优化与正样本相关的MSE而忽视整体MSE的增加,因为回归模型仍需保留区分非AMPs的能力。在这种任务设置中,我们认为一个好的回归模型应在不显著牺牲整体MSE的情况下,拥有较低的正样本相关指标值,如topK-MSE和pos-MSE。

在这一部分,作者通过一系列实验评估了SenseXAMP模型在AMPs分类和回归任务中的性能。以下是这一部分的简要描述:
预训练模型与传统PDs模型的互补性分析
为评估预训练模型(MPTM)和传统PDs模型(MPD)的互补性,作者在收集的不平衡分类数据集(包含7941个AMPs和28021个非AMPs)上进行了实验。结果表明,两种模型在错误样本上的重叠较少,表明它们在预测不同样本方面各有优势。因此,作者提出了融合预训练模型嵌入和PDs的合理方法,以解决单一输入模型无法解决的错误样本。
模型级集成分析
作者训练了一个XGBoost分类器,使用PDs作为输入,判断给定序列由哪种模型(MPTM或MPD)预测更准确。然而,XGBoost分类器的最终性能不理想,表明简单的模型级集成不足以显著提高整体预测性能。
与堆叠方法的比较
作者还进行了额外实验,通过堆叠方法集成两种不同模态的模型。结果表明,堆叠方法在提高灵敏度(Sn)方面有所帮助,但在其他指标上不如SenseXAMP。
与其他方法的比较
作者在多个数据集上比较了SenseXAMP与其他最新的AMP分类模型和回归模型。结果显示,SenseXAMP在综合指标(如准确率、MCC和F1分数)上显著优于其他模型,特别是在处理大规模和不平衡数据集时表现尤为突出。
特征级融合效果验证
通过对错误样本的详细分析,作者发现SenseXAMP能够准确分类单独使用预训练模型或PDs模型无法正确分类的样本。这表明,SenseXAMP有效地结合了多种模态的信息,显著提高了预测性能。
总结
实验结果表明,SenseXAMP在AMPs分类和回归任务中的性能优于现有的最先进模型,特别是在处理大规模和复杂数据集时。特征级融合方法展示了广泛的适用性,并避免了灾难性遗忘。
通过这些实验和结果,作者证明了SenseXAMP在AMPs任务中的有效性和优势,展示了其在实际应用中的巨大潜力。
论文复现
配置环境
1 | # clone project |
准备数据集
- ori_datasets
- esm_embeddings
- stc_info
- stc_datasets 注意:“stc_csv”数据集版本主要用于 SMEP
等比较方法,对于使用 SenseXAMP 不是必需的。 ## 下载model
checkpoints 按照README的步骤与给的脚本 结果报错,说找不到'./datasets/ori_datasets/AMPlify',我检查了我的目录结构,确实是和他给的一样的。 反思认为是./只能访问当前目录,返回上级目录应该是../,故修改,得之。 ## Generate esm-1b embeddings using our scripts 这一步的脚本根本就没写全,而且生成的AMPlify dataset有sequence缺失,所以我将ori_datasets的目标数据先写入一个txt文件,然后脚本读取这个文件,修改esm_emb_gen.py脚本如下
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23import os
import argparse
import pandas as pd
import sys
from structure_data_generate.cal_pep_des import cal_pep_fromlist
if __name__ == '__main__':
# generate structured data
data_dir = './datasets/ori_datasets/AMPlify'
files = os.listdir(data_dir)
out_dir = './datasets/stc_datasets/AMPlify'
os.makedirs(out_dir,exist_ok=True)
for file in files:
data_file = os.path.join(data_dir,file)
data = pd.read_csv(data_file, encoding="utf-8")
sequence = data['Sequence']
labels = data['Labels']
# labels = data['MIC']
peptides_list = sequence.values.copy().tolist()
out_path = os.path.join(out_dir,file)
print("output path: {}".format(out_path))
cal_pep_fromlist(peptides_list,output_path = out_path, labels=labels)## Run SenseXAMP ### 训练SenseXAMP1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77import argparse
import numpy as np
import pandas as pd
import torch
import h5py
import os
import esm_project as esm
from tqdm import tqdm
from typing import List
device = torch.device("cuda" if torch.cuda.is_available() else "cpu")
class EmbeddingProcessor:
"""
Generate esm embeddings for all proteins.
"""
def __init__(self) -> None:
self.pretrain_model, _ = esm.pretrained.esm1b_t33_650M_UR50S()
alphabet = esm.Alphabet.from_architecture("roberta_large")
self.batch_converter = alphabet.get_batch_converter()
self.pretrain_model = self.pretrain_model.to(device)
self.all_seqs = []
def get_seqs_from_list_file(self, seq_file: str):
"""
Get all sequences from a file
Args:
seq_file: path to file containing sequences
"""
with open(seq_file, 'r') as f:
self.all_seqs = [line.strip() for line in f.readlines()]
self.all_seqs = set(self.all_seqs)
def generate_embeddings(self, outdir, mode='all', fname='esm_embeddings.h5'):
"""
Generate embeddings for all sequence.
Args:
outdir: path to embedding file save
mode:
all or pooling.
fname: name of embedding file
"""
assert (mode == 'all') or (mode == 'pooling') or (mode == 'cls_token')
os.makedirs(outdir, exist_ok=True)
self.max_len = max(len(seq) for seq in self.all_seqs)
max_len = 64
if mode == 'all':
max_len = self.max_len
print("Max length: {}".format(self.max_len))
with h5py.File(os.path.join(outdir, fname), 'w') as hf:
for seq in tqdm(self.all_seqs):
data = [(seq, seq)]
_, _, batch_tokens = self.batch_converter(data, max_length=max_len)
batch_tokens = batch_tokens.to(device)
with torch.no_grad():
results = self.pretrain_model(batch_tokens, repr_layers=[33], return_contacts=True)
token_representations = results["representations"][33]
if mode == 'pooling':
embedding = token_representations.mean(1).squeeze(0) # [1280]
elif mode == 'cls_token':
embedding = token_representations[:, 0, :].squeeze(0) # cls token
else: # mode = 'all' (SenseXAMP use this type)
embedding = token_representations.squeeze(0) # [676,1280]
embedding = embedding.cpu().numpy()
hf.create_dataset(seq, data=embedding)
if __name__ == '__main__':
parser = argparse.ArgumentParser(description='A script to calculate esm_embeddings version of datasets')
parser.add_argument('--seq_file', default='C:/Users/LENOVO/Desktop/SenseXAMP/all_sequences.txt', help='path to file containing all sequences')
parser.add_argument('--fname', default='AMPlify.h5', help='name of output file name, name it xxx.h5')
args = parser.parse_args()
processor = EmbeddingProcessor()
processor.get_seqs_from_list_file(args.seq_file)
print(f"Number of sequences to embed: {len(processor.all_seqs)}")
processor.generate_embeddings('D:/SenseXAMP/datasets/esm_embeddings/all', mode='all', fname=args.fname)#### 在平衡分类数据集上训练1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67from ast import arguments
import os
import sys
import time
import random
import argparse
import torch
import numpy as np
# For DDP
import torch.distributed as dist
from torch.nn.parallel import DistributedDataParallel as DDP
from utils import Config,Logger
from Ampmm_base.runner import Runner
def parse_args():
parser = argparse.ArgumentParser(description='Training SenseXAMP benchmark')
parser.add_argument('--config', help='train config file path')
parser.add_argument('--mode', default='train', help='train or test')
# for ddp
parser.add_argument('--local_rank', type=int, default=0)
parser.add_argument('--seed', type=int, default=42, help='random seed')
args = parser.parse_args()
return args
def set_seed(seed_value=42):
"""
Set seed for reproducibility.
"""
random.seed(seed_value)
np.random.seed(seed_value)
torch.manual_seed(seed_value)
torch.cuda.manual_seed_all(seed_value)
# # for ddp
# parser.add_argument("--local_rank", default=-1, type=int)
if __name__ == '__main__':
args = parse_args()
local_rank = args.local_rank
# ddp init
torch.cuda.set_device(local_rank)
dist.init_process_group(backend='gloo')
# set random seed
cfg = Config.fromfile(args.config)
cfg.local_rank = args.local_rank
# create logger and work_dir
if dist.get_rank() == 0:
cfg.work_dir = os.path.join(cfg.work_dir,cfg.benchmark_name,cfg.dataset_name,args.mode,
time.strftime("%Y-%m-%d_%H-%M-%S", time.localtime()))
os.makedirs(cfg.work_dir,exist_ok=True)
logger = Logger(cfg.work_dir)
if args.local_rank == 0:
logger.info(args)
logger.info("Running with config:\n{}".format(cfg.text))
# set random seeds
if args.seed is not None:
logger.info('Set random seed to {}'.format(args.seed))
set_seed(args.seed)
runner = Runner(cfg,logger,args.local_rank,args.mode)
if args.mode == 'train':
runner.run()
elif args.mode == 'test':
runner.test()
else:
if args.local_rank == 0:
print("Please ensure args.mode to be train or test")
exit()训练结果:1
2CUDA_VISIBLE_DEVICES=0 python -m torch.distributed.launch --nproc_per_node 1 run.py \
--config ./configs/cls_task/benchmark_balanced_SenseXAMP.py --mode train#### 测试1
2[ 2024-07-10 11:44:20 ] Current best results:
{'TP': 0.36375078665827565, 'FP': 0.13971050975456262, 'TN': 0.36249213341724357, 'FN': 0.1340465701699182, 'Acc': 0.7262429200755192, 'Precision': 0.7225, 'Recall': 0.7307206068268015, 'Specificity': 0.7218045112781954, 'F1_score': 0.7265870521684474, 'MCC': 0.45253157041334263}1
2CUDA_VISIBLE_DEVICES=0 python -m torch.distributed.launch --nproc_per_node 1 run.py \
--config ./configs/cls_task/benchmark_balanced_SenseXAMP.py --mode test可视化测试结果1
2
3
4# Resume & Checkpoint setting
Resume = None # Resume from which ckpt to train
ckpt_path = 'C:/Users/LENOVO/Desktop/SenseXAMP/checkpoints/SenseXAMP_checkpoints/amp_cls/sensexamp_amplify.ckpt' # Checkpoint for test1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42import matplotlib.pyplot as plt
import numpy as np
import seaborn as sns
# 测试结果数据
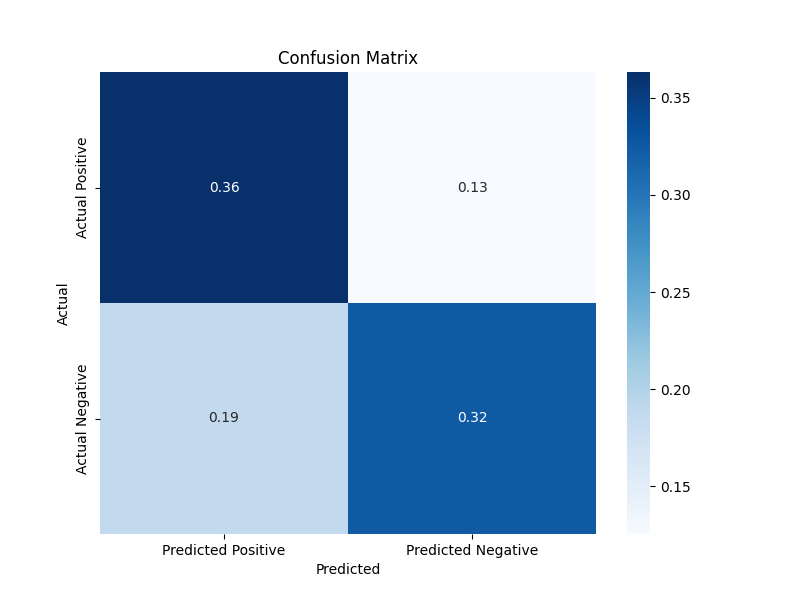
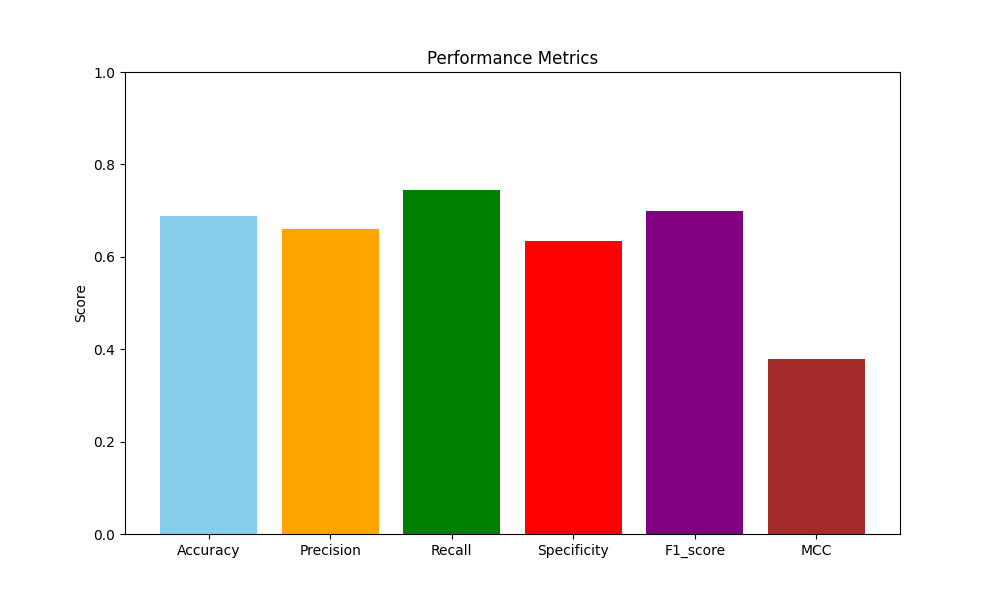
results = {
'TP': 0.3633501259445844,
'FP': 0.18702770780856423,
'TN': 0.3243073047858942,
'FN': 0.1253148614609572,
'Acc': 0.6876574307304786,
'Precision': 0.660183066361556,
'Recall': 0.7435567010309279,
'Specificity': 0.6342364532019704,
'F1_score': 0.6993939393939393,
'MCC': 0.37962791273329644
}
# 混淆矩阵
confusion_matrix = np.array([
[results['TP'], results['FN']],
[results['FP'], results['TN']]
])
# 绘制混淆矩阵
plt.figure(figsize=(8, 6))
sns.heatmap(confusion_matrix, annot=True, fmt=".2f", cmap="Blues", xticklabels=['Predicted Positive', 'Predicted Negative'], yticklabels=['Actual Positive', 'Actual Negative'])
plt.title("Confusion Matrix")
plt.xlabel("Predicted")
plt.ylabel("Actual")
plt.show()
# 绘制性能指标条形图
metrics = ['Accuracy', 'Precision', 'Recall', 'Specificity', 'F1_score', 'MCC']
values = [results['Acc'], results['Precision'], results['Recall'], results['Specificity'], results['F1_score'], results['MCC']]
plt.figure(figsize=(10, 6))
plt.bar(metrics, values, color=['skyblue', 'orange', 'green', 'red', 'purple', 'brown'])
plt.ylim(0, 1)
plt.title("Performance Metrics")
plt.ylabel("Score")
plt.show()